Plateforme de Transcriptomique Plante de Paris-Saclay, POPS
Présentation
La plateforme de transcriptomique des Plantes de Paris Saclay (POPS) propose depuis 2003 des services d’analyse du transcriptome des plantes à la communauté scientifique académique ou industrielle nationale ou internationale. Composée de techniciens, de biologistes, de bio-informaticiens et de statisticiens, elle est hébergée par l’Institut de Science des Plantes de Paris-Saclay sur le plateau de Saclay.
La plateforme POPS a pour mission de mutualiser des équipements, des compétences, des outils et des services afin de les proposer à une communauté scientifique publique ou privée, offrant ainsi une expertise forte et une offre intégrée dans le domaine de la transcriptomique végétale.
Les 3 axes de la plateforme transcriptomique sont :
- Service intégré à façon
- Développement
- Formation et animation scientifique
Elle est labellisée IBiSA et Infrastructure Scientifique Collective par INRAE. Elle fait partie du Réseau France Génomique et elle est certifiée ISO9001 depuis 2013.
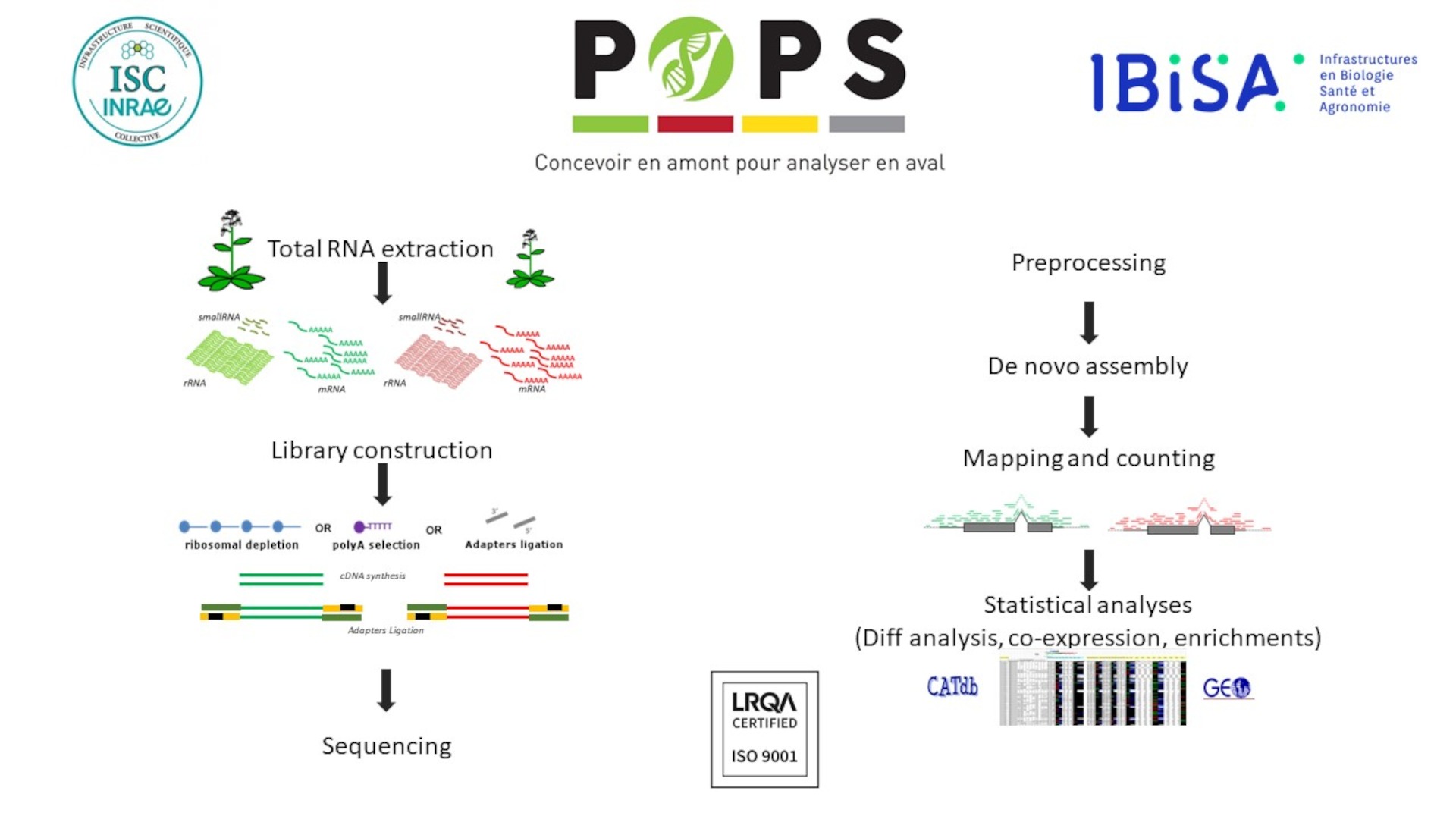
Activités
Expertises
La plateforme transcriptomique POPS réalise des projets d’étude du transcriptome en collaboration avec des équipes de recherche nationales et internationales. Elle offre une expertise pour l’analyse de l’expression comparative des gènes par l’utilisation du séquençage à haut débit de l’ARN, ou RNA-seq. Cette offre est complétée par l’analyse bio-informatique et statistique des données brutes permettant l’étude quantitative du transcriptome.
- Conception du projet en interaction avec le collaborateur (technologie, planification, …).
- Réalisation technique et production des données RNAseq.
- Analyses bio-informatiques et statistiques adaptées pour fournir des résultats exploitables.
- Interprétation des résultats.
- Prise en charge de la soumission dans les bases de données.
Points forts
- Versatilité : Diversité des organismes, Pluralité d’échantillons (type de culture, type d’organes, tissus …). Adaptabilité au nombre d’échantillons (test, moyen et haut débit).
- Interlocuteur dédié pour votre projet.
- Pipelines d’analyses bioinformatiques et statistiques développés spécifiquement pour nos applications.
Equipements
- bioanalyzer
- Fragment Analyzer
- Covaris
- Clariostar
- robot pipeteur Biomek FX
- Illumina NextSeq500
- Accès aux séquenceurs Illumina NovaSeq du CNS (Evry)
- Nanopore P2 solo
- cluster de calcul
Services proposés
Applications proposées
- Séquençage orienté des ARN polyAdenylés (le brin codant).
- Séquençage non orienté des ARN polyAdenylés.
- Séquençage des petits ARN : miRNA, siRNA …
- Séquençage de longs fragments (projets collaboratifs)
- Séquençage orienté des ARN non polyAdenylé (déplétion).
- Microtranscriptomique ou séquençage de très faible quantité d’ARN (à partir de 75pg d’ARN total).
- Métatranscriptomique ou séquençage du transcriptome de plusieurs espèces à partir d’un même échantillon.
- Empreintes.
- Cellule unique : en cours de développement.
Protocoles personnalisables en fonction de besoins spécifiques.
Options de séquençage
- Séquenceur NextSeq 500
- Accès aux séquenceurs HiSeq (CEA-CNS, Evry)
- Single Read ou Paired-End
- Longueurs de séquençage : 75, 100, 150 bases
Analyses bioinformatiques et statistiques
- Mapping
- Analyse différentielle de gènes
- Assemblage
- Traitement des smallRNA
Autres analyses possibles en collaboration avec l’équipe Réseaux Génomiques
Thématiques
Contact
Plateforme de Transcriptomique des Plantes de Paris-Saclay
Bâtiment 630, plateau du Moulon
Avenue des Sciences
91190 Gif-sur-Yvette
pops.ips2@universite-paris-saclay.fr
Site web
Tutelles
Université Paris Cité, Université Paris Saclay, Université Evry val d’Essonne, CNRS, INRAE
Unité de rattachement
Institut des Sciences des Plantes de Paris Saclay – UMR 9213
Réseaux
France Génomique
Labellisation
IBiSA, ISC
Certification
Norme ISO 9001
Plateformes dans la même thématique
Plateforme de spectroscopie et d’imagerie RPE
La plateforme appartient au Laboratoire de Chimie et Biochimie Pharmacologiques et Toxicologiques (UMR 8601), unité mixte de recherche CNRS/Université Paris Cité et est localisée sur le campus Saint Germain des Prés
IPS2 Epigenomic Platform (IPSEP)
In the recent years, the term « epigenetic » is rather used to define all the processes regulating gene expression and genome architecture, in addition to the information carried by the DNA sequence, that can be more transient or facultative in nature. Epigenetics information relies on DNA and protein modifications such as cytosine methylation (5mC, 5hmC) and histone post-translational modifications (e.g. acetylation, methylation). Many studies have highlighted the determining role of epigenetic regulations, not only in adaptation to stress and changing environments but also in the control of development. A better understanding of such processes provides the tools necessary to the development of innovative and efficient approaches to crop improvement. In this context IPSEP offers the solutions to any epigenomic and bioinformatic analysis you are seeking.
Plateau Imagerie Cellulaire et Cytométrie (PIC2) de BFA
Le plateau Imagerie cellulaire et cytométrie, créé en 2009 en même temps que l’unité BFA, donne accès à un équipement en imagerie cellulaire permettant l’observation dynamique de biomolécules dans des systèmes biologiques vivants
Ressource Parisienne en Bioinformatique Structurale (RPBS)
La plateforme Ressource Parisienne en Bioinformatique Structurale (RPBS) résulte de l’effort conjoint de plusieurs équipes de Bioinformatique Structurale de Paris pour promouvoir une ressource autour des thématiques de la Bioinformatique Structurale
Service de spectrométrie de Masse du LCBPT
Le service de spectrométrie de masse du Laboratoire de Chimie et Biochimie Pharmacologiques et Toxicologiques est une plateforme implantée au sein du Campus Saint Germain dont l’usage principal est la caractérisation et quantification de petites molécules organiques et/ou de petites biomolécules (nucléotides, peptides). Cette plateforme est dotée de trois spectromètres de masse couplés à de la chromatographie liquide et un spectromètre couplé à de la chromatographie en phase gazeuse. L’ensemble de ces matériels est géré par un assistant ingénieur, Patrice Gerardo, qui effectue un peu plus de 10 000 analyses de routine par an. Il assure également la mise au point et le développement de techniques et méthodes analytiques nouvelles.
Analyses cellulaires multidimensionnelles de l’UAR3612 P-MIM
La plateforme Analyses Cellulaires Multidimensionnelles est une plateforme qui permet l’analyse des cellules à différents niveaux : expressions et activité de protéines à la surface de, dans et par la cellule. Elle appartient à l’Unité de service “Plateformes Mutualisées de l’Institut du Médicament” labellisée US25 Inserm, UAR 3612 CNRS, située à la Faculté de Pharmacie de Paris, à proximité immédiate des nombreux centres de recherche de Paris. Ouverte à la communauté scientifique académique et privée, elle met son expertise et ses équipements au service des chercheurs.
