Plateforme « InterATOME »
Présentation
La plateforme InterATOME est intégrée à l’équipe de recherche OGE – Expression génomique des organites au sein du Département Interactions Plantes Microorganismes et Réseaux de l’Institut des Sciences des Plantes – Paris-Saclay (IPS2) sur le plateau de Saclay.
La plateforme est dédiée au criblage à haut-débit des interactions protéine-protéine par un système de Double-Hybride sur levures (Y2H). Elle offre ses services dans le cadre de collaborations nationales et internationales.
Activités
Expertises
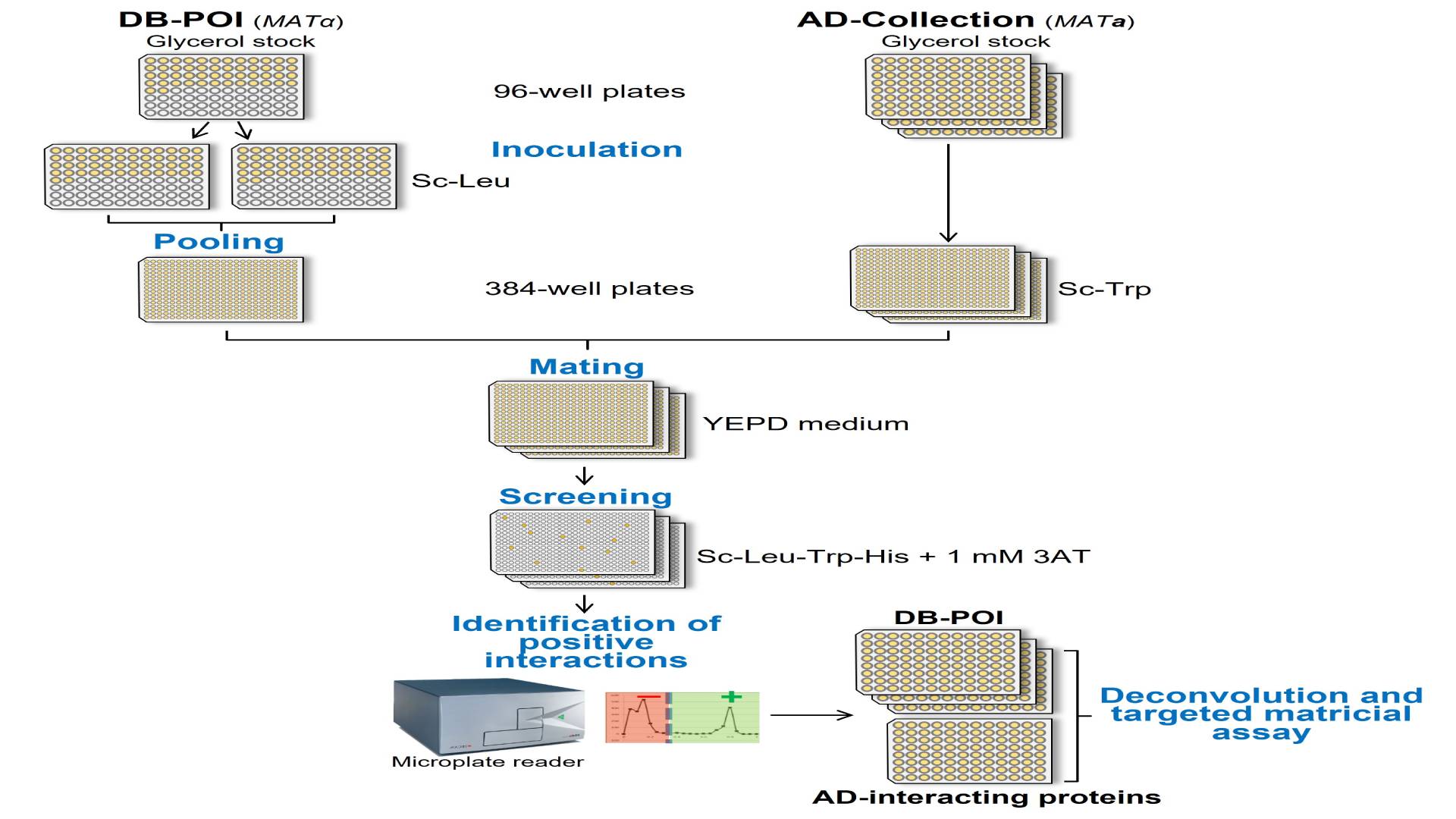
Grâce à l’utilisation de plaques 384 puits et à un protocole entièrement en milieu liquide, la plate-forme InterATOME est capable de cribler un pool de 50 protéines hybrides (DB- X) contre la collection de protéines hybrides AD-AIM (environ 12000 protéines d’Arabidopsis) en deux mois. Les plasmides portant les ORFs codant pour les protéines d’intérêt fournis par nos collaborateurs dans le vecteur pDEST – DB employé sur la plateforme sont utilisés pour transformer des levures puis les protéines DB-X hybrides sont testées pour éliminer celles capables d’autoactivation. Les levures exprimant les protéines DB-X hybrides sont cultivées individuellement dans des milieux sélectifs, puis combinées en pools de 50 clones et criblées contre la banque AD-AIM. Après identification des protéines candidates, une analyse matricielle en Y2H est effectuée par « déconvolution » des 50 DB-proies et par tests individuels contre les candidats AD-appâts. Une étape finale de séquençage des protéines AD- et DB- permet la validation de l’identité des protéines en interaction.
Equipements
La plateforme est équipée de plusieurs robots nécessaires à la manipulation de plaques :
- Qpix2XT Colony picker (Genetix). Le QPix combine une analyse d’image intelligente avec une automatisation précise pour un criblage rapide et efficace de grandes collections. Le QPix peut prélever 3 000 colonies par heure et automatiser la réplication des plaques, cherry-picking, l’étalement, etc.
- Orbitor RS Benchtop Microplate mover (ThermoFisher-Scientific). L’Orbitor Microplate Mover est un déplaceur de microplaques de paillasse compact. Son bras télescopique bidirectionnel permet d’avoir un espace de travail étendu à 360° tout en assurant une grande précision de manipulation.
- Multidrop 384 Microplate dispenser (ThermoFisher-Scientific). Le Multidrop 384 est un distributeur automatisé de microplaques conçu pour le criblage à haut débit et la distribution de microvolumes dans des plaques de 384 et 96 puits.
Services proposés
La plateforme InterATOME propose une expertise allant de la conception du projet en interaction avec le collaborateur aux analyses des données avec la prise en charge de la soumission dans les bases de données. La plateforme est aussi très impliquée dans l’animation scientifique : Journées dédiées aux analyses omiques végétales et visites des installations. Plus de détails à l’adresse https://ips2.u-psud.fr/fr/plateformes/spomics-interatome-metabolome-transcriptome/interatome-plateforme-interactomique.html
Thématiques
Contact
Plateforme InterATOME le plateforme d’interactomique de l’IPS2
IPS2 Bâtiment 630
Plateau de Moulon, avenue des Sciences
91192 Gif-sur-yvette
spomics-interactome@ips2.upsaclay.fr
Site web
Tutelles
Université Paris-Saclay, Université Paris-Cité, Université Evry, CNRS, INRAE
Unité de rattachement
Institut des Sciences des Plantes de Paris Saclay – UMR 9213
Plateformes dans la même thématique
Plateforme de criblage par ARN interférence (PARi)
La plateforme PARi est dédiée au criblage à haut débit (High-Throughput Screening, HTS) et à haut contenu (High-Content Screening, HCS) de banques de perturbateurs biologiques (siRNAs, petites molécules…) sur tests cellulaires.
Plateforme Protéomique SFR Necker (PPN) de la SFR Necker
Fondée en 2006, la plateforme PPN fait partie de la SFR Necker et du réseau des plateformes protéomiques de l’Université Paris Cité. Au cœur d’un centre hospitalo-universitaire d’excellence, elle offre une technologie et une expertise de pointe pour mieux comprendre les maladies
Plateforme de Verrerie Scientifique et Technique de l’UFR de chimie
Au sein de cette plateforme technique sont produits des prototypes, des pièces uniques ou de petites séries de fabrication spéciale pour les unités de recherche qui utilisent le verre et ses dérivés dans leurs travaux.
Plateforme Histologie, Immunomarquage, Microdissection laser (Hist’IM) de l’Institut Cochin
La plateforme HistIM est ouverte aux équipes de l’institut Cochin mais aussi aux équipes extérieures, institutionnelles ou privées.
Elle propose des prestations techniques d’anatomie et pathologie, animale et humaine.
Plateforme Proteomique Structurale et Fonctionnelle (ProtéoSeine@IJM)
ProteoSeine@IJM est la plateforme protéomique de l’Institut Jacques Monod, UMR 7592 Université de Paris Cité – CNRS.
Les activités de la plateforme incluent du service analytique, de la recherche et du développement technique et méthodologique ainsi qu’une veille technologique active.
Plateforme Génomique de l’Institut Imagine – SFR Necker
La plateforme génomique d’Imagine a pour mission d’offrir des prestations de haut niveau dans le domaine du séquençage à haut débit (NGS) depuis 2010 et de l’expression génique à la communauté scientifique d’Imagine et du site Necker-Enfants Malades principalement mais pas exclusivement
Cordeliers Genotyping & Biochemistry (CGB) du CRC
Le CGB (Centre de Génotypage et de Biochimie) du Centre de Recherche des Cordeliers (CRC) assure une prestation de service en génotypage et la mise à disposition d’appareils et matériel de biologie moléculaire et de biochimie. Ses infrastructures, ses équipements et le support scientifique et technique de son personnel sont à la disposition de tous les membres du CRC, de ses Universités de tutelle (Université Paris Cité et Sorbonne Université) ainsi qu’aux autres utilisateurs académiques ou privés. Les activités de génotypage du CGB sont engagées dans une démarche qualité depuis 2013 et sont certifiées iso 9001:v2015 et NFX-50-900. Le CGB est sous la responsabilité d’un responsable technique et opérationnel, Hermine Kakanakou.
IPS2 Epigenomic Platform (IPSEP)
In the recent years, the term « epigenetic » is rather used to define all the processes regulating gene expression and genome architecture, in addition to the information carried by the DNA sequence, that can be more transient or facultative in nature. Epigenetics information relies on DNA and protein modifications such as cytosine methylation (5mC, 5hmC) and histone post-translational modifications (e.g. acetylation, methylation). Many studies have highlighted the determining role of epigenetic regulations, not only in adaptation to stress and changing environments but also in the control of development. A better understanding of such processes provides the tools necessary to the development of innovative and efficient approaches to crop improvement. In this context IPSEP offers the solutions to any epigenomic and bioinformatic analysis you are seeking.
