Plateforme de Transcriptomique Plante de Paris-Saclay, POPS
Présentation
La plateforme de transcriptomique des Plantes de Paris Saclay (POPS) propose depuis 2003 des services d’analyse du transcriptome des plantes à la communauté scientifique académique ou industrielle nationale ou internationale. Composée de techniciens, de biologistes, de bio-informaticiens et de statisticiens, elle est hébergée par l’Institut de Science des Plantes de Paris-Saclay sur le plateau de Saclay.
La plateforme POPS a pour mission de mutualiser des équipements, des compétences, des outils et des services afin de les proposer à une communauté scientifique publique ou privée, offrant ainsi une expertise forte et une offre intégrée dans le domaine de la transcriptomique végétale.
Les 3 axes de la plateforme transcriptomique sont :
- Service intégré à façon
- Développement
- Formation et animation scientifique
Elle est labellisée IBiSA et Infrastructure Scientifique Collective par INRAE. Elle fait partie du Réseau France Génomique et elle est certifiée ISO9001 depuis 2013.
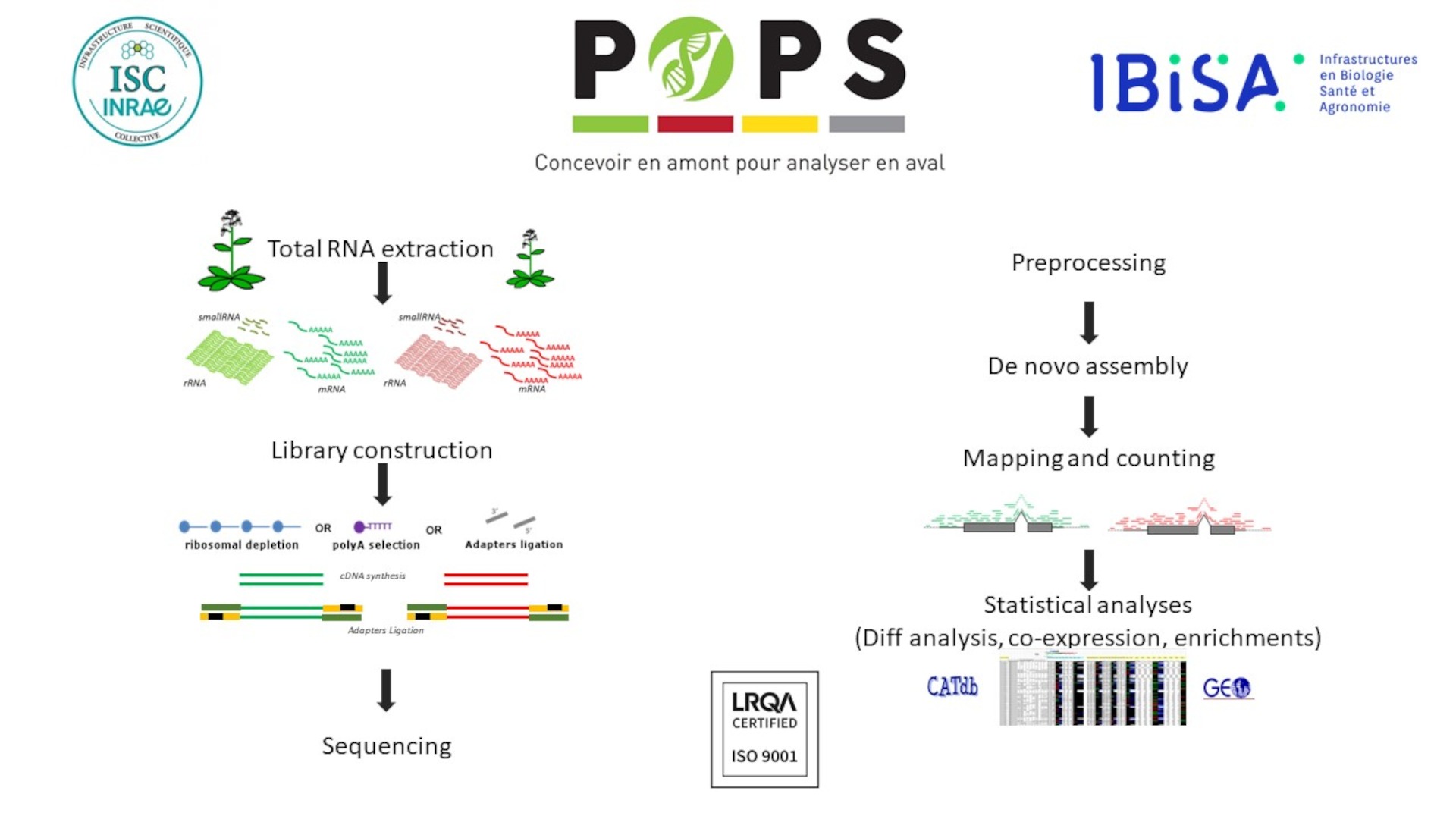
Activités
Expertises
La plateforme transcriptomique POPS réalise des projets d’étude du transcriptome en collaboration avec des équipes de recherche nationales et internationales. Elle offre une expertise pour l’analyse de l’expression comparative des gènes par l’utilisation du séquençage à haut débit de l’ARN, ou RNA-seq. Cette offre est complétée par l’analyse bio-informatique et statistique des données brutes permettant l’étude quantitative du transcriptome.
- Conception du projet en interaction avec le collaborateur (technologie, planification, …).
- Réalisation technique et production des données RNAseq.
- Analyses bio-informatiques et statistiques adaptées pour fournir des résultats exploitables.
- Interprétation des résultats.
- Prise en charge de la soumission dans les bases de données.
Points forts
- Versatilité : Diversité des organismes, Pluralité d’échantillons (type de culture, type d’organes, tissus …). Adaptabilité au nombre d’échantillons (test, moyen et haut débit).
- Interlocuteur dédié pour votre projet.
- Pipelines d’analyses bioinformatiques et statistiques développés spécifiquement pour nos applications.
Equipements
- bioanalyzer
- Fragment Analyzer
- Covaris
- Clariostar
- robot pipeteur Biomek FX
- Illumina NextSeq500
- Accès aux séquenceurs Illumina NovaSeq du CNS (Evry)
- Nanopore P2 solo
- cluster de calcul
Services proposés
Applications proposées
- Séquençage orienté des ARN polyAdenylés (le brin codant).
- Séquençage non orienté des ARN polyAdenylés.
- Séquençage des petits ARN : miRNA, siRNA …
- Séquençage de longs fragments (projets collaboratifs)
- Séquençage orienté des ARN non polyAdenylé (déplétion).
- Microtranscriptomique ou séquençage de très faible quantité d’ARN (à partir de 75pg d’ARN total).
- Métatranscriptomique ou séquençage du transcriptome de plusieurs espèces à partir d’un même échantillon.
- Empreintes.
- Cellule unique : en cours de développement.
Protocoles personnalisables en fonction de besoins spécifiques.
Options de séquençage
- Séquenceur NextSeq 500
- Accès aux séquenceurs HiSeq (CEA-CNS, Evry)
- Single Read ou Paired-End
- Longueurs de séquençage : 75, 100, 150 bases
Analyses bioinformatiques et statistiques
- Mapping
- Analyse différentielle de gènes
- Assemblage
- Traitement des smallRNA
Autres analyses possibles en collaboration avec l’équipe Réseaux Génomiques
Thématiques
Contact
Plateforme de Transcriptomique des Plantes de Paris-Saclay
Bâtiment 630, plateau du Moulon
Avenue des Sciences
91190 Gif-sur-Yvette
pops.ips2@universite-paris-saclay.fr
Site web
Tutelles
Université Paris Cité, Université Paris Saclay, Université Evry val d’Essonne, CNRS, INRAE
Unité de rattachement
Institut des Sciences des Plantes de Paris Saclay – UMR 9213
Réseaux
France Génomique
Labellisation
IBiSA, ISC
Certification
Norme ISO 9001
Plateformes dans la même thématique
Cordeliers Histology, Imaging, Cytometry & Spatial-Omics (CHICS) du CRC
Le Centre d’Histologie, d’Imagerie et de Cytométrie (CHIC) du Centre de Recherche des Cordeliers (CRC) offre des équipements et des services dans les domaines de l’histologie, de la biologie spatiale, de l’imagerie et de la cytométrie en flux. Ses infrastructures, ses équipements et le support scientifique et technique de son personnel sont à la disposition de tous les membres du CRC, de ses Universités de tutelle (Université Paris Cité et Sorbonne Université) ainsi qu’aux autres utilisateurs académiques ou privés.
Plateforme d’Analyses Moléculaires et Structurales (PAMS)
La plateforme d’Analyses Moléculaires et Structurales (AMS) propose ses services aux laboratoires de recherche académique et industrielle.
NeurImag de l’IPNP
Bienvenue sur la plate-forme d’imagerie de l’Institut de Psychiatrie et Neurosciences de Paris, localisée dans le 14ième arrondissement de Paris près du parc Montsouris. La plateforme évolue en collaboration directe avec l’INSERM, l’Université Paris Cité et l’Hôpital Sainte-Anne. La plateforme NeurImag est labélisée IBISA et membre de l’Infrastructure France Bio Imaging. Elle fait également partie du consortium UP3 des plateformes photoniques de l’université de Paris Cité.
Plateforme Proteomique Structurale et Fonctionnelle (ProtéoSeine@IJM)
ProteoSeine@IJM est la plateforme protéomique de l’Institut Jacques Monod, UMR 7592 Université de Paris Cité – CNRS.
Les activités de la plateforme incluent du service analytique, de la recherche et du développement technique et méthodologique ainsi qu’une veille technologique active.
Plateforme Génomique de l’Institut Imagine – SFR Necker
La plateforme génomique d’Imagine a pour mission d’offrir des prestations de haut niveau dans le domaine du séquençage à haut débit (NGS) depuis 2010 et de l’expression génique à la communauté scientifique d’Imagine et du site Necker-Enfants Malades principalement mais pas exclusivement
Plateforme « InterATOME »
La plateforme InterATOME est intégrée à l’équipe de recherche OGE – Expression génomique des organites au sein du Département Interactions Plantes Microorganismes et Réseaux de l’Institut des Sciences des Plantes – Paris-Saclay (IPS2) sur le plateau de Saclay.
La plateforme est dédiée au criblage à haut-débit des interactions protéine-protéine par un système de Double-Hybride sur levures (Y2H). Elle offre ses services dans le cadre de collaborations nationales et internationales.
Cordeliers Genotyping & Biochemistry (CGB) du CRC
Le CGB (Centre de Génotypage et de Biochimie) du Centre de Recherche des Cordeliers (CRC) assure une prestation de service en génotypage et la mise à disposition d’appareils et matériel de biologie moléculaire et de biochimie. Ses infrastructures, ses équipements et le support scientifique et technique de son personnel sont à la disposition de tous les membres du CRC, de ses Universités de tutelle (Université Paris Cité et Sorbonne Université) ainsi qu’aux autres utilisateurs académiques ou privés. Les activités de génotypage du CGB sont engagées dans une démarche qualité depuis 2013 et sont certifiées iso 9001:v2015 et NFX-50-900. Le CGB est sous la responsabilité d’un responsable technique et opérationnel, Hermine Kakanakou.
Plateforme de Biochimie du CRI
La plateforme de Biochimie & métabolisme du petit animal associée au Centre d’Explorations Fonctionnelles Intégré (CRI, UMR 1149, http://www.cri1149.fr/les-plateformes/), UFR de Médecine à Université Paris Cité existe depuis 1999.
