Plateforme de Transcriptomique Plante de Paris-Saclay, POPS
Présentation
La plateforme de transcriptomique des Plantes de Paris Saclay (POPS) propose depuis 2003 des services d’analyse du transcriptome des plantes à la communauté scientifique académique ou industrielle nationale ou internationale. Composée de techniciens, de biologistes, de bio-informaticiens et de statisticiens, elle est hébergée par l’Institut de Science des Plantes de Paris-Saclay sur le plateau de Saclay.
La plateforme POPS a pour mission de mutualiser des équipements, des compétences, des outils et des services afin de les proposer à une communauté scientifique publique ou privée, offrant ainsi une expertise forte et une offre intégrée dans le domaine de la transcriptomique végétale.
Les 3 axes de la plateforme transcriptomique sont :
- Service intégré à façon
- Développement
- Formation et animation scientifique
Elle est labellisée IBiSA et Infrastructure Scientifique Collective par INRAE. Elle fait partie du Réseau France Génomique et elle est certifiée ISO9001 depuis 2013.
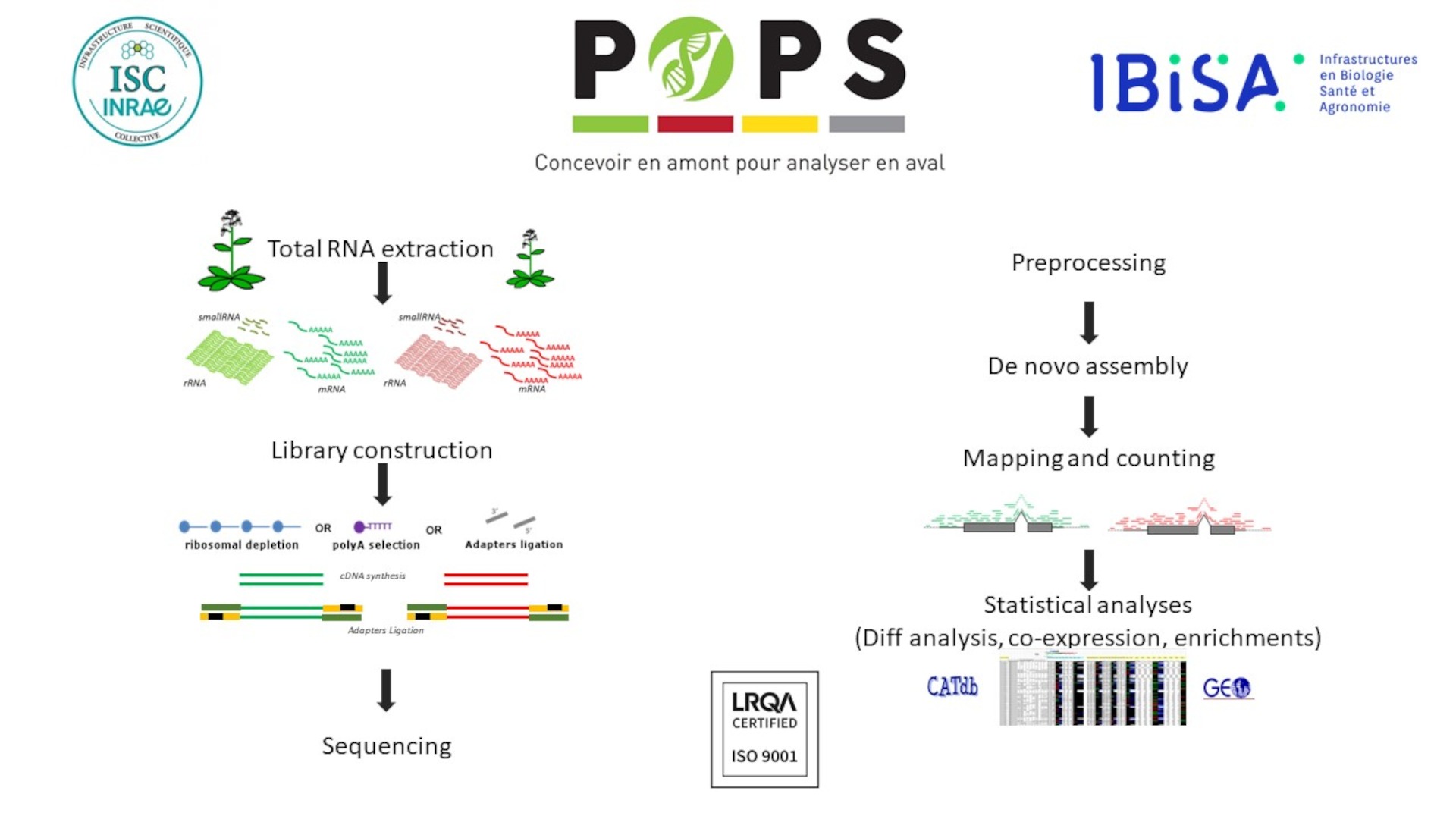
Activités
Expertises
La plateforme transcriptomique POPS réalise des projets d’étude du transcriptome en collaboration avec des équipes de recherche nationales et internationales. Elle offre une expertise pour l’analyse de l’expression comparative des gènes par l’utilisation du séquençage à haut débit de l’ARN, ou RNA-seq. Cette offre est complétée par l’analyse bio-informatique et statistique des données brutes permettant l’étude quantitative du transcriptome.
- Conception du projet en interaction avec le collaborateur (technologie, planification, …).
- Réalisation technique et production des données RNAseq.
- Analyses bio-informatiques et statistiques adaptées pour fournir des résultats exploitables.
- Interprétation des résultats.
- Prise en charge de la soumission dans les bases de données.
Points forts
- Versatilité : Diversité des organismes, Pluralité d’échantillons (type de culture, type d’organes, tissus …). Adaptabilité au nombre d’échantillons (test, moyen et haut débit).
- Interlocuteur dédié pour votre projet.
- Pipelines d’analyses bioinformatiques et statistiques développés spécifiquement pour nos applications.
Equipements
- bioanalyzer
- Fragment Analyzer
- Covaris
- Clariostar
- robot pipeteur Biomek FX
- Illumina NextSeq500
- Accès aux séquenceurs Illumina NovaSeq du CNS (Evry)
- Nanopore P2 solo
- cluster de calcul
Services proposés
Applications proposées
- Séquençage orienté des ARN polyAdenylés (le brin codant).
- Séquençage non orienté des ARN polyAdenylés.
- Séquençage des petits ARN : miRNA, siRNA …
- Séquençage de longs fragments (projets collaboratifs)
- Séquençage orienté des ARN non polyAdenylé (déplétion).
- Microtranscriptomique ou séquençage de très faible quantité d’ARN (à partir de 75pg d’ARN total).
- Métatranscriptomique ou séquençage du transcriptome de plusieurs espèces à partir d’un même échantillon.
- Empreintes.
- Cellule unique : en cours de développement.
Protocoles personnalisables en fonction de besoins spécifiques.
Options de séquençage
- Séquenceur NextSeq 500
- Accès aux séquenceurs HiSeq (CEA-CNS, Evry)
- Single Read ou Paired-End
- Longueurs de séquençage : 75, 100, 150 bases
Analyses bioinformatiques et statistiques
- Mapping
- Analyse différentielle de gènes
- Assemblage
- Traitement des smallRNA
Autres analyses possibles en collaboration avec l’équipe Réseaux Génomiques
Thématiques
Contact
Plateforme de Transcriptomique des Plantes de Paris-Saclay
Bâtiment 630, plateau du Moulon
Avenue des Sciences
91190 Gif-sur-Yvette
pops.ips2@universite-paris-saclay.fr
Site web
Tutelles
Université Paris Cité, Université Paris Saclay, Université Evry val d’Essonne, CNRS, INRAE
Unité de rattachement
Institut des Sciences des Plantes de Paris Saclay – UMR 9213
Réseaux
France Génomique
Labellisation
IBiSA, ISC
Certification
Norme ISO 9001
Plateformes dans la même thématique
Plateforme d’irradiation de l’UMR-E 008
L’institut de radiobiologie cellulaire et moléculaire (iRCM) du CEA (UMR-E 008)dispose d’une plateforme d’irradiation ouverte aux laboratoires souhaitant étudier les effets des rayonnements ionisants sur le vivant et sur la santé humaine.
Plateforme Organoides Cérébraux Humains et Neurodéveloppement HumBO (Human Brain Organoids) de NeuroDiderot
HumBO est une Plateforme Inserm et Université de Paris Cité dédiée à la modélisation du neurodéveloppement normal et pathologique au moyen d’organoïdes cérébraux humains.
Plateforme technologique de l’IRSL
L’IRSL (Institut de Recherche Saint-Louis, Université Paris-Cité, Hôpital Saint-Louis, Paris 10 ) est une structure de recherche de l’Université Paris-Cité adossée à l’hôpital Saint-Louis, regroupant et hébergeant les unités mixtes de recherche INSERM,CNRS, UP, CEAEA, APHP.
Epigénomique Fonctionnelle de l’Epigénétique & Destin cellulaire
La plateforme Epigénomique Fonctionnelle propose à la communauté scientifique une expertise dans le domaine de l’épigénétique
Genome Engineering in Epigenetics (GENIE)
Le plateau technique « Genome ENgeneering In Epigenetics » ( GENIE ) est axée sur les diverses applications des technologies CRISPR/Cas dans le domaine de l’épigénétique
Plateau d’analyse structurale et fonctionnelle du globule rouge
Le plateau d’analyse structurale et fonctionnelle du globule rouge fédère les expertises des équipes de l’unité Biologie Intégrée du Globule Rouge (BIGR) et celles de certaines équipes du labex GR-Ex comme celle du Pr Olivier Hermine
Plateforme de Calcul Haute-Performance NOVA
Nova vise à répondre aux besoins de calculs haute-perfomance des membres d’Université Paris Cité
Plateforme de Modélisation des Molécules, Matériaux et Biomolécules – P3MB
La Plateforme de Modélisation des Molécules, Matériaux et Biomolécules (P3MB) est gérée par le laboratoire ITODYS (CNRS UMR7086) au sein de l’équipe Chimie Théorique et Modélisation
