enSCORE (engineered Spinal and Cortical Organoid coRE)
Présentation
La plateforme enSCORE a été créée en 2021 pour apporter un support à un consortium interdisciplinaire de chercheurs qui s’intéressent au développement embryonnaire du système nerveux central et ses dérégulations par des mutations génétiques retrouvées dans des pathologies infantiles. Elle vise à faciliter l’accès aux chercheurs à des organoïdes neuraux dérivés de cellules souches pluripotentes induites humaines (iPSC). Ces organoïdes sont les seuls modèles du développement prénatal du cerveau et de la moelle épinière chez l’homme. Ils présentent aussi l’avantage de permettre une exploration du développement en combinant des approches de génétique, de biologie cellulaire et moléculaire et de pharmacologie.
La plateforme est financée par le Labex Université Paris Cité « Who am I ? » dans le cadre du projet transversal (Organoïdes neuraux pour étudier les interactions entre les signaux mécaniques et transcriptionnels qui sous-tendent le neurodéveloppement normal et pathologique).
Activités
Expertises de la plateforme enSCORE
L’optimisation, la standardisation et la caractérisation de cultures d’iPSC humaines
La plateforme a mis en place et offre un soutien et une formation à la culture de d’iPSC humaines ainsi qu’une caractérisation de la qualité de ces cellules (absence de mycoplasmes, marqueurs de pluripotence, intégrité chromosomique, potentiel de différenciation).
Le design et la génération de lignées transgéniques par édition du génome
En interaction avec les membres des équipes intéressées et du plateau GENIE (http://parisepigenetics.com/gen/), la plateforme met en place des procédures de modélisations de pathologies, en reproduisant les mutations présentes chez les patients par des approches d’édition du génome avec les systèmes CRISPR/Cas dans des lignées d’iPSC.
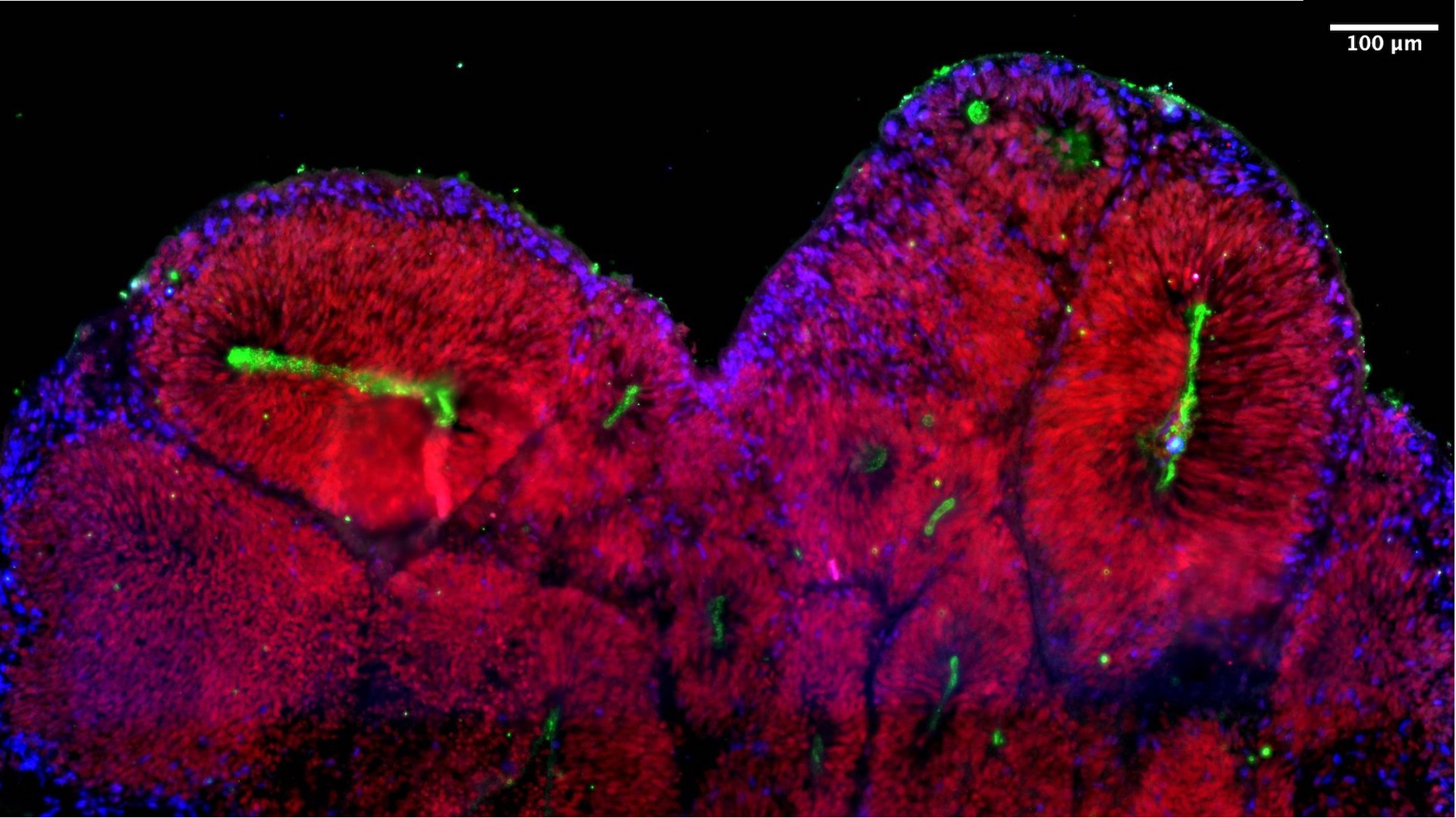
La production et la caractérisation d’organoïdes neuraux (cérébraux et spinaux)
La plateforme emploie des protocoles variés (fournisseurs, publiés, ou mis au point par des collaborateurs) pour générer les organoïdes cérébraux et spinaux et forme les utilisateurs. Les organoïdes générés sont phénotypés par analyses de coupes fines au cryostat et marquage par immunofluorescence ainsi que par cytométrie en flux (Plateforme Imagoseine).”
Équipements
La plateforme dispose d’une pièce de culture cellulaire de confinement L2 complètement équipée pour la culture des iPSC et la différentiation des organoïdes.
- Deux PSM réservables en ligne ;
- Deux incubateurs CO2.
Les équipements spécifiques comprennent notamment :
- Une loupe binoculaire (Dynascope Lynx, Vision Engineering) placée sous PSM permettant l’observation et la manipulation stérile des cultures. Cette loupe est équipée d’un système vidéo relié à un écran permettant la prise de photos/vidéos et permet la formation aisée des utilisateurs de la plateforme ;
- Un agitateur orbital (Dutscher) nécessaire à la différentiation des organoïdes cérébraux est également installé dans l’un des incubateurs.
Les membres de la plateforme et ses utilisateurs bénéficient également de l’ensemble des équipements de l’Institut Jacques Monod notamment :
- Un cryostat Leica CM3050S (datant de 2004)
Après formation par Kamal Bouhali, Mikaelle Bocel ou Vanessa Ribes ce système est réservable en ligne - Un système de transfection (Nucleofector 4D) permettant la nucléofection des lignées iPSC et la génération des lignées transgéniques.
Après formation par Kamal Bouhali, ce système est réservable en ligne.
Services proposés
La plateforme propose une formation et un accompagnement des équipes de recherches sur :
- La culture des iPSC et le contrôle qualité de ces cultures ;
- Le développement de stratégies d’édition du génome (en collaboration avec la plateforme GENIE (http://parisepigenetics.com/gen/) ;
- La différentiation en organoïdes cérébraux et spinaux ainsi que la caractérisation phénotypique de ces organoïdes ;
- Elle accueille également des stagiaires de différentes formations (BTS, Master).
Thématiques
Contact
enSCORE
Bâtiment Buffon B, 3ème étage, salle 342B
15 rue Hélène Brion
75013, Paris
enscore@services.cnrs.fr
Site web
Tutelles
Université Paris Cité, CNRS, Inserm
Unités de rattachement
Epigénétique et Destin Cellulaire – UMR 7216, Institut Cochin – UMR 8104, Institut Jacques Monod – UMR 7592
Plateformes dans la même thématique
Plateforme d’Imagerie Cellulaire de la SFR Necker
La plateforme d’imagerie cellulaire de la SFR Necker est spécialisée dans la visualisation et l’analyse de la structure et des processus dynamiques, au niveau cellulaire et tissulaire jusqu’au niveau de l’organisme entier. Sa mission est de fournir aux chercheurs des instruments d’optiques de pointe leur permettant de répondre à leurs problématiques scientifiques et de développer des méthodes d’imagerie nouvelles et toujours plus innovantes. La plateforme est ouverte à tous les instituts de recherche et aux industries privées.
Plateforme Génomique de l’Institut Imagine – SFR Necker
La plateforme génomique d’Imagine a pour mission d’offrir des prestations de haut niveau dans le domaine du séquençage à haut débit (NGS) depuis 2010 et de l’expression génique à la communauté scientifique d’Imagine et du site Necker-Enfants Malades principalement mais pas exclusivement
Plateforme – Imagerie Cellulaire et Moléculaire (PICMO)
PICMO (Plateforme d’Imagerie Cellulaire et MOléculaire) est une plateforme de microscopie optique et électronique
Analyse du métabolisme chez le petit animal : Métabol’IC de l’Institut Cochin
La vocation de la plateforme Métabol’IC est d’offrir aux scientifiques une caractérisation exhaustive des composantes de l’homéostasie énergétique de la cellule au petit animal
Plateforme « InterATOME »
La plateforme InterATOME est intégrée à l’équipe de recherche OGE – Expression génomique des organites au sein du Département Interactions Plantes Microorganismes et Réseaux de l’Institut des Sciences des Plantes – Paris-Saclay (IPS2) sur le plateau de Saclay.
La plateforme est dédiée au criblage à haut-débit des interactions protéine-protéine par un système de Double-Hybride sur levures (Y2H). Elle offre ses services dans le cadre de collaborations nationales et internationales.
Cordeliers Genotyping & Biochemistry (CGB) du CRC
Le CGB (Centre de Génotypage et de Biochimie) du Centre de Recherche des Cordeliers (CRC) assure une prestation de service en génotypage et la mise à disposition d’appareils et matériel de biologie moléculaire et de biochimie. Ses infrastructures, ses équipements et le support scientifique et technique de son personnel sont à la disposition de tous les membres du CRC, de ses Universités de tutelle (Université Paris Cité et Sorbonne Université) ainsi qu’aux autres utilisateurs académiques ou privés. Les activités de génotypage du CGB sont engagées dans une démarche qualité depuis 2013 et sont certifiées iso 9001:v2015 et NFX-50-900. Le CGB est sous la responsabilité d’un responsable technique et opérationnel, Hermine Kakanakou.
Plateforme de Biochimie du CRI
La plateforme de Biochimie & métabolisme du petit animal associée au Centre d’Explorations Fonctionnelles Intégré (CRI, UMR 1149, http://www.cri1149.fr/les-plateformes/), UFR de Médecine à Université Paris Cité existe depuis 1999.
Plateforme de spectroscopie et d’imagerie RPE
La plateforme appartient au Laboratoire de Chimie et Biochimie Pharmacologiques et Toxicologiques (UMR 8601), unité mixte de recherche CNRS/Université Paris Cité et est localisée sur le campus Saint Germain des Prés
