Plateforme d’imagerie moléculaire (iMAP) du CRI
Présentation
La plateforme iMAP appartient à l’Inserm, UMR 1149, Centre de Recherche sur l’Inflammation (CRI). Elle est adossée à l’équipe de recherche « De l’inflammation au cancer dans les maladies digestives » située sur le site de l’hôpital Beaujon (Clichy la Garenne). iMAP est membre d’un réseau structuré de plateformes protéomiques identifiés par l’Université de Paris et regroupant Proteom’IC 3P5 (Institut Cochin, E-F Gautier – F. Guillonneau), PPN (SFR Necker, C Guerrera), et ProteoSeine (Institut J Monod, JM Camadro – G. Chevreux), qui développent des approches protéomiques complémentaires performantes pour l’analyse et la quantification des protéines.
Etant ouverte à toute la communauté scientifique académique ou industrielle, iMAP est donc engagée dans une démarche qualité (ISO 9001) afin d’assurer à ses utilisateurs : traçabilité des échantillons et suivi du projet.
Activités
Expertises
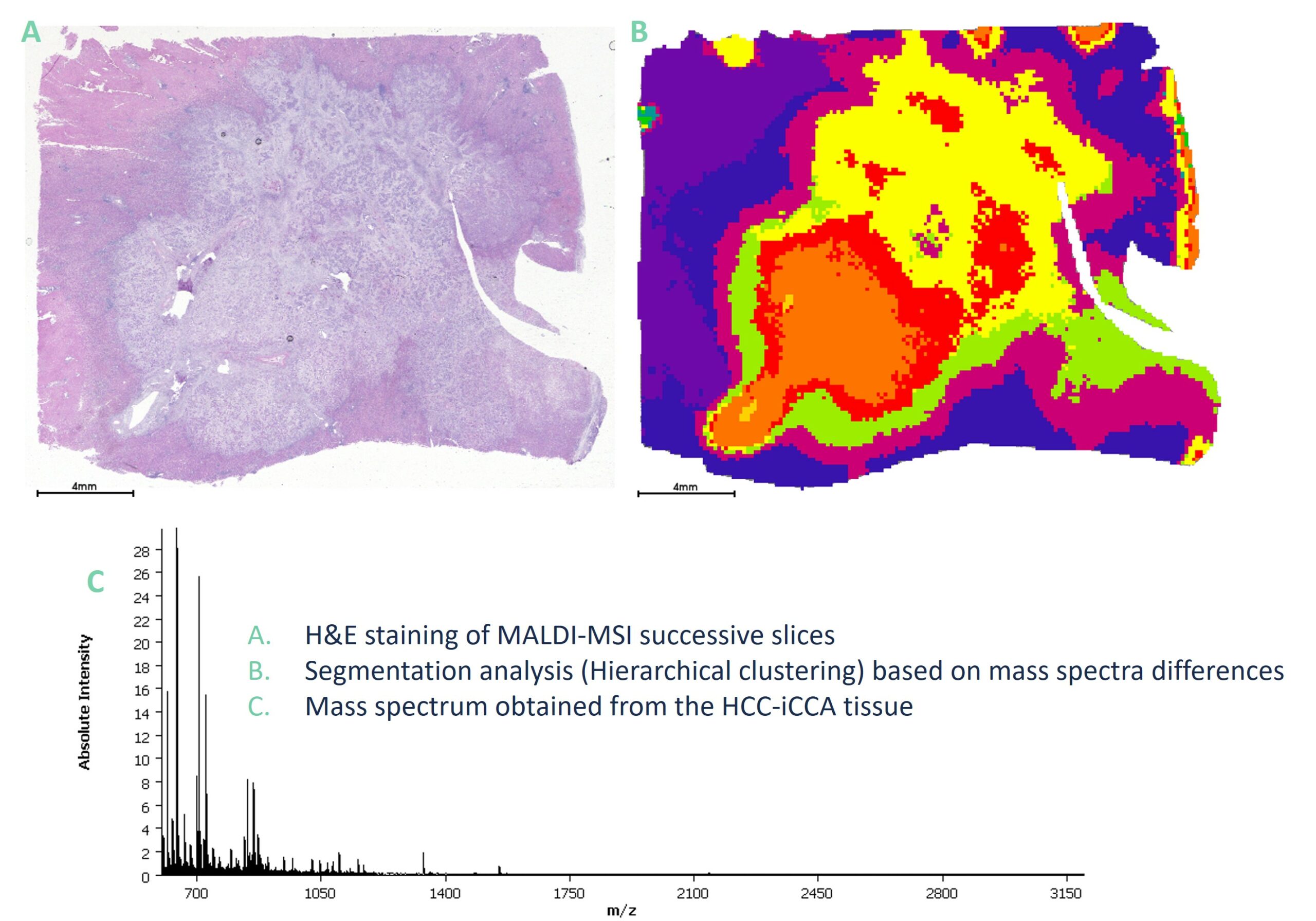
iMAP possède une expertise en imagerie par spectrométrie de masse sur tissus FFPE (fixés en paraffine) et sur tissus congelés. Vous pourrez donc réaliser des analyses protéomiques, lipidomique et glycomique sur :
- des coupes entières, des biopsies ou de TMAs (Tissue MicroArray) issus de blocs FFPE ;
- des coupes issus d’échantillons congelés ;
- des échantillons humains ou animaux ;
- tout type d’organes.
Grâce à ces collaborations il vous sera possible de:
- réaliser de l’imagerie multimodale en combinant l’imagerie MALDI avec les analyses histologiques et/ou immunohistochimiques (Service d’anatomie-pathologie de l’hôpital Beaujon) ;
- réaliser des traitements en bio-informatique sur mesure.
Équipements
Nous disposons du matériel nécessaire à la réalisation des coupes tissulaires (cryomicrotome, microtome), au prétraitement de vos lames (lavage, déparaffinage, démasquage antigénique …) aux acquisitions des données par spectrométrie de masse (MALDI TOF-TOF) ainsi qu’au traitement statistique des données.
Nos équipements :
- Un mMicrotome s (Leica) ;
- Un Cryostats (Leica) ;
- Une balance de précision ;
- Un spectromètre de masse type MALDI-TOF RapifleX (Bruker Daltonics) ;
- Un sprayeur de matrice TM-sprayer (HTX) ;
- Une chambre de démasquage antigénique (Biocare Medical, EUROBIO ABCYS) ;
- Un système pour dépôt de la matrice par sublimation (assemblage fait maison avec l’aide de Bruker Daltonics ®) ;
- Un scanner de lames Plustek (Aperio) ;
- Des logiciels pour le traitement des données d’imagerie MALDI : FlexImaging 4.1 et Scils Lab 2023a Pro ;
- Congélateur -80°C pour la conservation des échantillons ;
- Congélateur -20°C pour la conservation des réactifs.
Services proposés
iMAP propose des prestations d’imagerie par spectrométrie de masse in situ. Nous réalisons différentes analyses Omics : Protéomiques, Glycomique, Lipidomique, sur du tissu congelé ou FFPE. Les échantillons seront déposés sur des lames ITO (Indium Tin Oxide) 2,5×7,5 cm. La plateforme réalise principalement des cartographies moléculaires afin d’étudier la biodistribution de molécules cibles. Notre prestation démarre de la découpe de vos échantillons et se poursuit jusqu’à l’analyse des données.. Nous pouvons vous accompagner pour la mise en forme de vos données et la rédaction de publications associées.
En parallèle, iMAP réalise des colorations histologiques et immunohistochimique dans le but de proposer de l’imagerie multimodale. Des protocoles de microdissection peuvent également être réaliser lors de collaboration afin de procéder par la suite à des analyses biochimiques ou couplées à de la LC/MS (Liquid Chromatography Mass Spectrometry).
Thématiques
Contact
iMAP Pavillon Abrami
Hopital Beaujon
100 boulevard du Général Leclerc
92110, Clichy-la-Garenne
plateforme.imap@gmail.com
Site web
Tutelles
Université Paris Cité, Inserm, CNRS
Unités de rattachement
Centre de Recherche sur l’Inflammation – UMR-S 1149
Plateformes dans la même thématique
Plateau technique de biologie cellulaire en hypoxie du B3OA
PrésentationLe plateau technique de biologie cellulaire en hypoxie du B3OA est situé sur le site Villemin.Il permet la culture de cellules et la réalisation d’expériences à des concentrations en...
Plateforme LUMIERE (L’Unité Materno-foetale en Imagerie Echographique et Résonance magnétiquE)
La Plateforme LUMIERE (L’Unité Materno-fœtale en Imagerie Échographique & Résonance magnétiquE) créée au sein de l’Hôpital Necker – Enfants Malades, est une plateforme de recherche clinique unique au monde, dédiée à l’imagerie médicale de la femme enceinte, du fœtus et du placenta.
Plateforme technologique de l’IRSL
L’IRSL (Institut de Recherche Saint-Louis, Université Paris-Cité, Hôpital Saint-Louis, Paris 10 ) est une structure de recherche de l’Université Paris-Cité adossée à l’hôpital Saint-Louis, regroupant et hébergeant les unités mixtes de recherche INSERM,CNRS, UP, CEAEA, APHP.
Plateforme de microscopie Photonique IRCM
La plateforme de microscopie photonique de l’Institut de Radiobiologie Cellulaire et Moléculaire (iRCM) met à la disposition des chercheurs les équipements nécessaires et les compétences techniques en microscopie photonique pour tout projet scientifique en sciences du vivant utilisant les techniques d’imagerie. Le personnel fournit une assistance et une formation à la microscopie photonique, à l’acquisition et au traitement d’image.
Epigénomique Fonctionnelle de l’Epigénétique & Destin cellulaire
La plateforme Epigénomique Fonctionnelle propose à la communauté scientifique une expertise dans le domaine de l’épigénétique
Plateau d’analyse structurale et fonctionnelle du globule rouge
Le plateau d’analyse structurale et fonctionnelle du globule rouge fédère les expertises des équipes de l’unité Biologie Intégrée du Globule Rouge (BIGR) et celles de certaines équipes du labex GR-Ex comme celle du Pr Olivier Hermine
Plateforme de Calcul Haute-Performance NOVA
Nova vise à répondre aux besoins de calculs haute-perfomance des membres d’Université Paris Cité
Plateforme de Modélisation des Molécules, Matériaux et Biomolécules – P3MB
La Plateforme de Modélisation des Molécules, Matériaux et Biomolécules (P3MB) est gérée par le laboratoire ITODYS (CNRS UMR7086) au sein de l’équipe Chimie Théorique et Modélisation